Rで外れ値検定 | グラブス検定を行い箱髭図と蜂群図を描く
データに大きく外れた値がある時、実験系を見直すべきか?あるいは、明らかなヒューマンエラーは無かったか? と考える必要があるのは勿論ですが、今あるデータから適切な基準で外れ値を除外して議論したいという場面もあるかと思います。
本記事は、R言語でoutlierパッケージのグラブス検定(grubbs test)を使用し、外れ値を判断する方法についての記事です。
0.スミノルフ・グラブスの外れ値検定とは?
原理
この論文に書いてあるのですが、統計の素人である私には詳細は理解できませんでした。
F. E. Grubbs, Procedures for Detecting Outlying Observations in Samples, Technometrics
11,
1-21 (1969).
私が理解できた範囲では、以下のような内容でした。
・最大値と最小値、平均値から離れているのはどっち?
・その値をグラブスが考えた式で計算し、グラブス検定量という「外れ具合」を算出する。
・「外れ具合」がグラブスが考えた基準値より大きかったら、それは外れ値。
・論文中に、「例えばn=7で有意水準を5%とするなら基準値はこの値」という対応表が載っている。
論文に使って大丈夫か?
ご自身の研究分野の先行研究で使われているか?が1つの基準になると思います。私は、がんに関する研究をしており、自分と似たような実験手法の文献で「Grrubs検定によって外れ値を決定し、白丸で示した」という旨の記述がある文献を2報見つけたので、ボスに相談した上で使うことにしました。
G. Samaranayake, et al., Thioredoxin-1 protects against androgen receptor-induced redox
vulnerability in castration-resistant prostate cancer, Nature Commun. 8, 1204
(2017).
S. Kato, et al., P38 pathway as a key downstream signal of connective tissue growth factor to
regulate metastatic potential in non-small-cell lung cancer, Cancer Sci. 107,
1416-1421
(2016).
1.SCV形式のデータを用意する
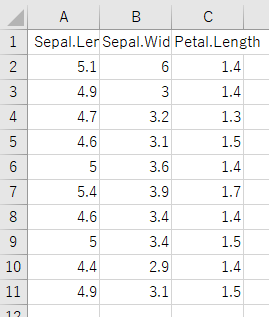
よく例として使われるデータセットであるirisを一部改変したデータを使用します。Excelにコピペしてください。
| Sepal.Length | Sepal.Width | Petal.Length |
| 5.1 | 6 | 1.4 |
| 4.9 | 3 | 1.4 |
| 4.7 | 3.2 | 1.3 |
| 4.6 | 3.1 | 1.5 |
| 5 | 3.6 | 1.4 |
| 5.4 | 3.9 | 1.7 |
| 4.6 | 3.4 | 1.4 |
| 5 | 3.4 | 1.5 |
| 4.4 | 2.9 | 1.4 |
| 4.9 | 3.1 | 1.5 |
↓Excelにコピペして、
名前をつけて保存→csv形式で保存。
2.Rでoutlierパッケージをインストール
初回のみ行う作業
R studioのコンソール画面に以下をコピペして、Enterを押す。
install.packages("outliers")
package ‘outliers’ successfully unpackedみたいなことを言われたらOKです。最初の頃はインストールが上手くいかなかったのですが、「R パッケージ インストールできない」でググって調べた通りにしたらできました。
R studioを再起動する度に必要な作業
R studioのコンソール画面に以下をコピペして、Enterを押す。今度はクオテーションマーク(" ")は無しでOKです。
library(outliers)
何も起こらなくて不安ですが、それで大丈夫です。
念のため確認すると、Packagesの一覧にチェックが入っているはずです。なお、library(outliers)→Enterの代わりに、ここにチェックを入れても同じことなのでそれでもOKです。
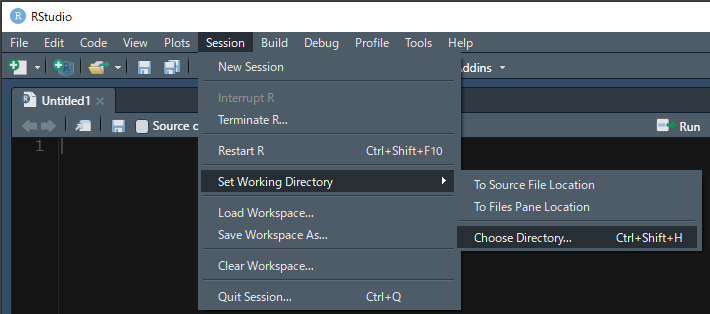
3.ディレクトリを移動する
R studioの"session"→"Set working Directory"→"Choose Directory"でCSVファイルを保存した場所を開く。
ショートカットキー:Ctrl + Shift + H
コンソールにsetwd("ディレクトリ名")と打ち込んでも良いのですが、windowsの場合、全ての逆スラッシュを手動で2本にする必要があります。
4.コードをコピペして実行
以下のコードをコピペし、2行目を読み込みたいファイル名に変えて実行。
しかし、全てのカラム(列)に外れ値検定を行いたくない場合は、以下の変更が必要です。・3行目を「test.all <- FALSE」に変更。
・14行目を変更する。例えばデータが4列あって、2列目だけは行いたくない場合は「test.collum <- c(TRUE, FALSE, TRUE, TRUE)」とする。
remove(list=ls())
imported.filename <- "testdata.csv" #読み込みたいファイル名に変える
test.all <- TRUE #全部のカラムに外れ値検定を行うならTRUE、そうでなければFALSE
#どのカラムに対して検定を行うか?の情報を配列text.collumに格納する
imported.table <- read.csv(imported.filename)
test.collum <- logical(ncol(imported.table))
if (test.all == TRUE) {
for (i in 1:ncol(imported.table)) {
test.collum[i] <- TRUE
i <- i + 1
}
} else {
test.collum <- c(FALSE, TRUE, TRUE) #行いたくないカラムをFALSEにする
}
#gurubbs.testを実行した結果を、配列result[]とp.value[]に格納する。この時点では文字列型
result <- character(ncol(imported.table))
p.value <- numeric(ncol(imported.table))
i <- 1
for (i in 1:ncol(imported.table)) {
result.temp <- grubbs.test(imported.table[[i]])
result[i] <- result.temp[[2]]
p.value[i] <- result.temp[[3]]
i <- i + 1
}
#今後の処理を行いやすくするため、外れ値が何行目かをまとめておく
#resultは文字列型なので、数値型の配列outlier[]を作って格納しておく
outlier.row <- numeric(ncol(imported.table))
outlier <- numeric(ncol(imported.table))
i <- 1
for (i in 1:ncol(imported.table)) {
#grep()は文字列検索して存在したらTRUEを返す
#外れ値が最大値の場合はgrep()で1が返るため、if関数の引数が1になる
#外れ値が最小値の場合はgrep()で長さ0のベクトルが返るため、if関数の引数が0になる
#max()中のna.rm=TRUEは欠損値を除外するオプション
if (length(grep("highest", result[i]))) {
outlier.row[i] <- which.max(imported.table[[i]])
outlier[i] <- max(imported.table[[i]], na.rm = TRUE)
} else {
outlier.row[i] <- which.min(imported.table[[i]])
outlier[i] <- min(imported.table[[i]], na.rm = TRUE)
}
}
#p.valueが0.05未満かどうかの情報を、配列p.value.lowに格納する
p.value.low <- logical(ncol(imported.table))
i <- 1
for (i in 1:ncol(imported.table)) {
p.value.low[i] <- p.value[i] < 0.05
i <- i + 1
}
#除外したくない列として手動で指定したカラムがあれば反映する
exclude.ok <- logical(ncol(imported.table))
if (test.all == TRUE) {
exclude.ok <- p.value.low
} else {
exclude.ok <- p.value.low & test.collum
}
#外れ値を除外した表を作る
excluded.table <- imported.table
i <- 1
for (i in 1:ncol(imported.table)) {
if (exclude.ok[i]) {
excluded.table[outlier.row[i], i] <- NA
}
i <- i + 1
}
View(excluded.table)
#外れ値を格納するための表を作っておく
outlier.table <- imported.table
i <- 1
for (i in 1:ncol(imported.table)) {
outlier.table[i] <- NA
i <- i + 1
}
#outlier.tableに外れ値を格納する
i <- 1
for (i in 1:ncol(imported.table)) {
if (exclude.ok[i]) {
outlier.table[outlier.row[i], i] <- outlier[i]
}
i <- i + 1
}
#result.tableに結果をまとめて表示する
result.table <- rbind(result, outlier, p.value, outlier.row, p.value.low, test.collum, exclude.ok)
colnames(result.table) <- colnames(imported.table)
View(result.table)
result.tableのタブをクリックすると、片側グラブス検定の結果と、p値が表示されます。1番上の行が空欄の場合は、outlierパッケージが正常に機能していません。インストールができているか、library(outliers)をやり忘れてないか確認してください。
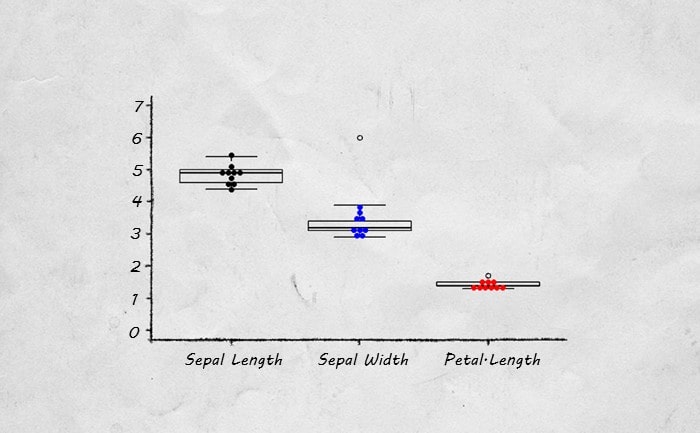
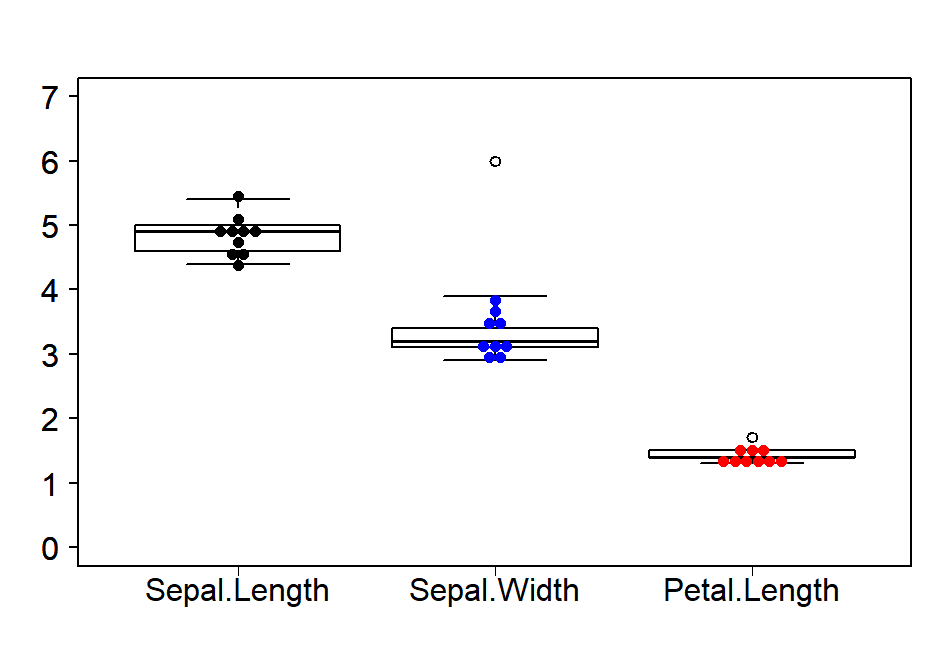
excluded.tableのタブをクリックすると、外れ値が除外された表が表示されます。このコードでは、片側グラブス検定でp<0.05の値を外れ値としました。
5.箱ひげ図に蜂群図を重ねて描画する
beeswarmというパッケージが必要なため、まずは以下の2行を順番に実行。「2.Rでoutlierパッケージをインストール」に書いた通り、install.packageをするのは初回のみで大丈夫です。
install.packages("beeswarm")library(beeswarm)以下のコードをコピペし、実行するとグラフが出ます。
「4.コードをコピペして実行」を実行したときに「excluded.table」と「outlier.table」がR studioに記憶され、それを描画します。そのため、「4.コードをコピペして実行」の直後に実行する必要があります。
1行目のwin.graphと、8行目のyMaxの値はデータに合わせて変えてください。
win.graph(10, 7) #プロットエリアのサイズ、単位はインチ
#win.graph()が無いとRStudio右下にグラフが表示され、縦横サイズを保存前に聞かれる
par(tcl = -0.5, #目盛の長さ:負の数だと外向き
las = 1, #目盛の数字の向き
lwd = 2, #線の太さ
cex.axis=2 #文字の大きさ
)
yMax <- 7 #y軸の範囲
box.color <- "white" #多色にする場合はc("","","")と表記
beeswarm.color <- c("black", "blue", "red")
#これより下は変更する必要無し
boxplot(excluded.table,
range = 0,#0は外れ値無しという指定
ylim = c(-0.01,yMax), #最小値が0だとプロットが潰れてしまう
medlwd = 3, #中央値の太さ
col = box.color
)
beeswarm(excluded.table,
method = "center",
col = beeswarm.color,
pch = 16, #プロットの種類:16は黒丸
cex = 1.5, #プロットの大きさ
ylim = c(-0.01,yMax),
add = TRUE #グラフを上書きする
)
beeswarm(outlier.table,#外れ値を描画する
pch = 1, #プロットの種類:1は白丸
cex = 1.5, #プロットの大きさ
ylim = c(-0.01,yMax),
add = TRUE #グラフを上書きする
)
axis(side = 2,lwd = 2) #目盛線の太さを上書きする,side2は左
今回のコードでは、このようなグラフが描けます。
以上、グラブス検定で外れ値を除外してグラフを描画する方法でした。